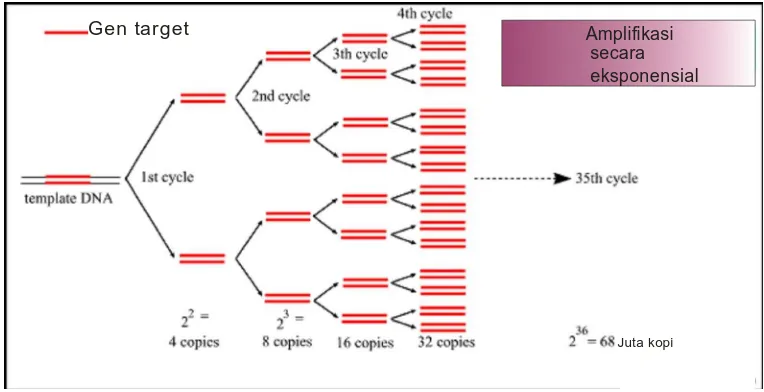
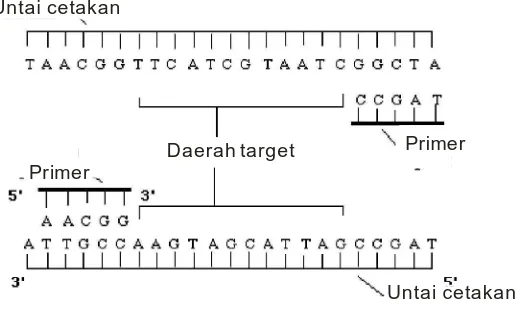
Polymerase Chain Reaction.
Teks penuh
Gambar
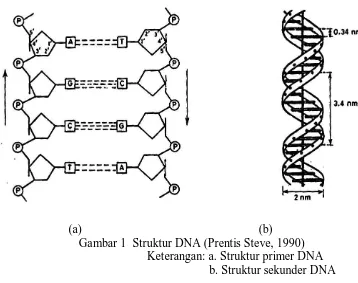
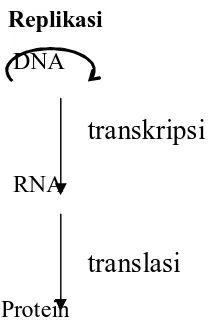
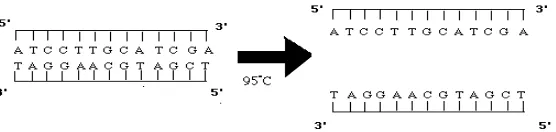
Garis besar
Dokumen terkait
IAIN Syekh Nurjati Cirebon menerapkan Kurikulum Berbasis Kompetensi (KBK) dengan tujuan agar lulusan memiliki kompetensi yang menjadi tujuan dan sasaran jurusan/ prodi.. Mata
Dengan menggunakan metode efficient sequencing yang menerapkan algoritma Fuzzy C-Means clustering dan K-Means clustering pada hybrid recommender system diharapkan dapat..
Tes dan konseling adalah pintu gerbang utama menuju pengobatan denga ARV yang merupakan bagian dari pelayanan kesehatan paripurna bagi ODHA dengan pendekatan
Pembangunan jalan alternatif kota Idi bertujuan untuk menghindari masalah kemacetan di sekitar kota idi, pembangunan jalan tersebut telah dimulai sejak tahun 2010 dengan
Dalam konteks sistem pendidikan di Malaysia, di antara kemahiran generik yang dikenalpasti perlu dimiliki oleh pelajar adalah kemahiran berkomunikasi, kemahiran menggunakan
Hasil analisis ragam menunjukkan bahwa penambahan rafinosa dengan dosis yang berbeda dalam pengencer susu skim terhadap persentase motilitas sapi Ongole menunjukkan
Hasil penelitian menunjukkan pengelolaan sumber daya udang dogol di perairan samboja dapat dilakukan dengan dua kondisi pengelolaan optimal yaitu Maximum Sustainable Yield