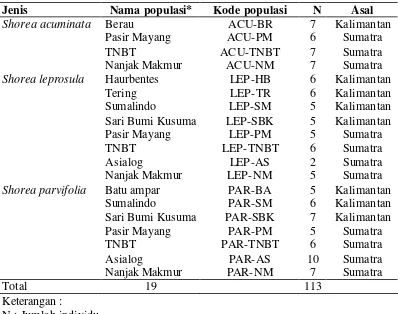
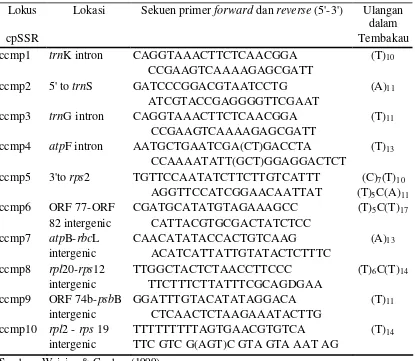
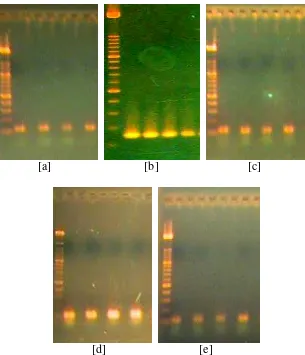
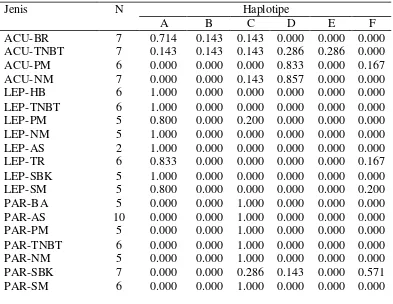
Variasi DNA Kloroplas Shorea spp (Shorea acuminata Dyer, Shorea leprosula Miq dan Shorea parvifolia Dyer) Berdasarkan Penanda Mikrosatelit
Bebas
54
0
0
Teks penuh
Gambar
+7
Dokumen terkait