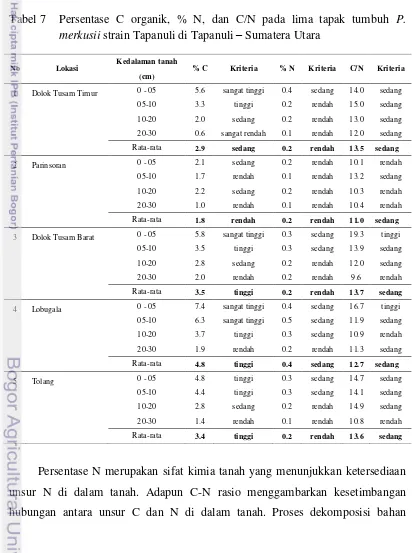
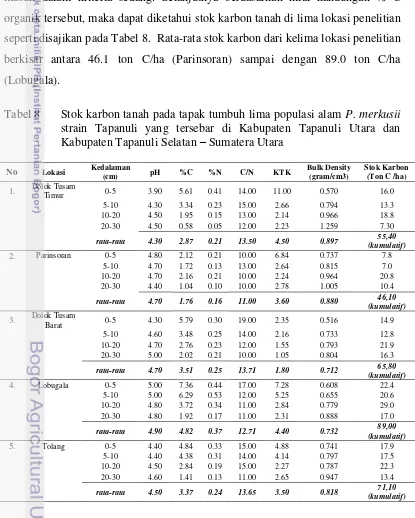
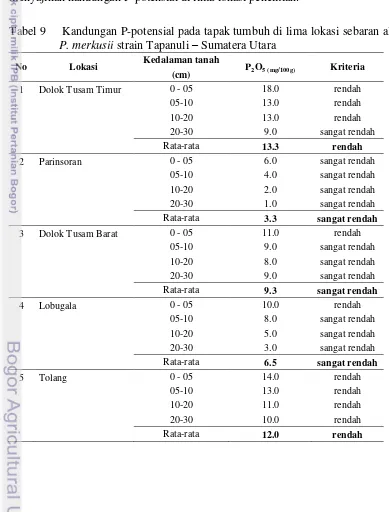
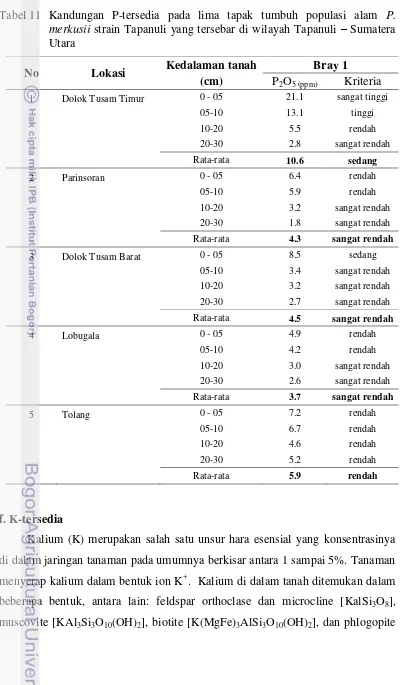
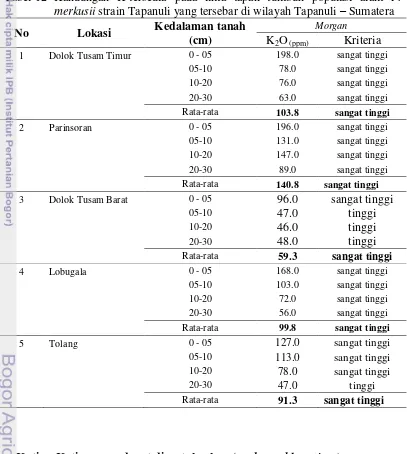
Analysis of land cover changes, genetic structure, and carbon biomass stock of pinus Merkusii Jungh Et De Vriese Strain Tapanuli In Its Natural Distribution In North Sumatra
Teks penuh
Gambar
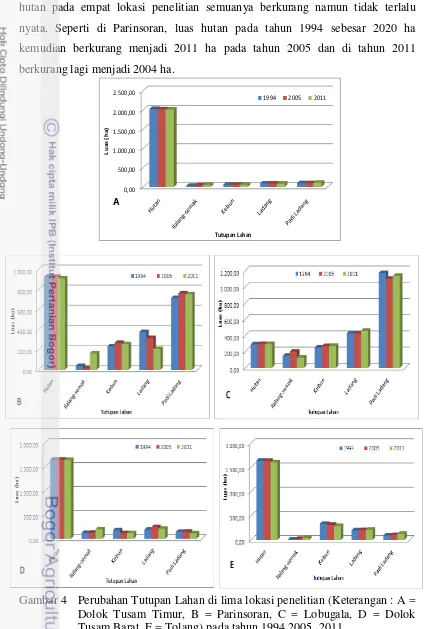
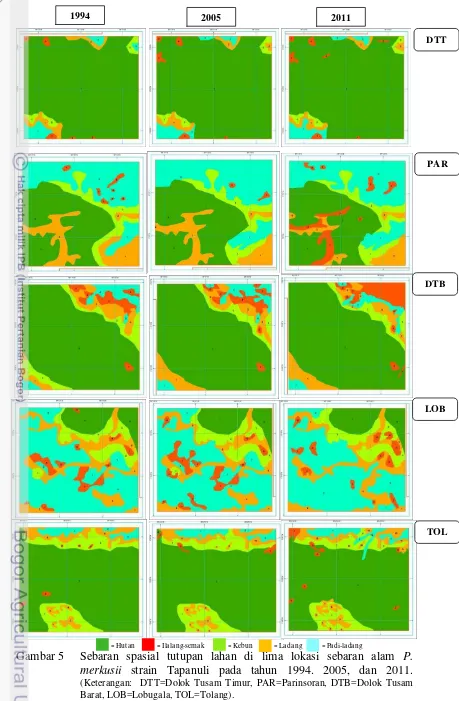
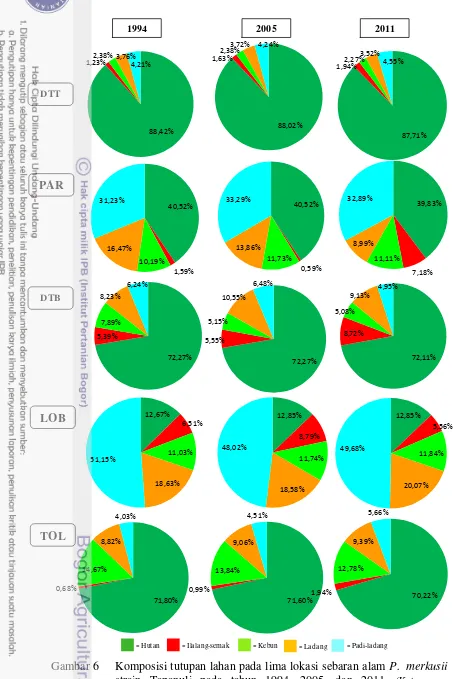
Dokumen terkait
Sehubungan dengan telah dilakukannya evaluasi administrasi, teknis dan kewajaran harga serta formulir isian Dokumen Kualifikasi untuk penawaran paket pekerjaan tersebut diatas,
Berdasarkan hasil penelitian pada model regresi diketahui bahwa variabel struktur modal berpengaruh negatif dan secara statistik signifikan terhadap nilai
memahami strategi dan teknik bimbingan dan konseling; jenis-jenis layanand. bimbingan dan konseling; dasar-dasar pemahaman peserta didik;
Ditinjau dari diskriminan tersebut, maka persamaan kuadrat dapat dibagi menjadi tiga macam, yaitu :.. D > 0 : Mempunyai dua akar real
Proses mengentry data muatan barang Database barang XOR /\ /\ Posisi muatan barang valid Posisi muatan barang valid Posisi muatan barang tidak valid Data posisi muatan barang
Demikian Berita Acara Pemberian Penjelasan (BAPP) pekerjaan ini dibuat dengan penuh rasa tanggung jawab dan untuk dipergunakan sebagaimana mestinya. Unit Layanan
[r]
Kerusakan berat yang sering terjadi pada basicmeter adalah putusnya kawat halus di dalam kumparan yang terletak di dalam kotak. Akibatnya basicmeter tidak